KAIST, 뇌 구조 정확히 볼 수 있는 3차원 분석기술 개발
- 김태진 기자
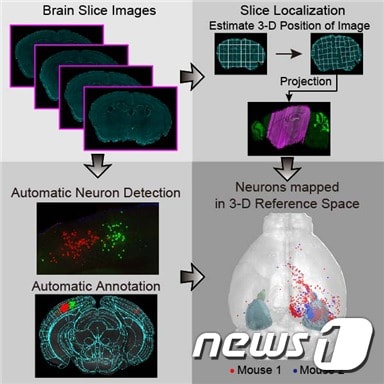
(대전=뉴스1) 김태진 기자 = 국내 연구진이 뇌 구조를 정확히 볼 수 있는 3차원 분석기술을 개발했다.
KAIST(총장 신성철)는 바이오및뇌공학과 백세범 교수 연구팀이 뇌신경과학 연구에서 광범위하게 사용되는 실험용 쥐의 뇌 절편 영상을 자동으로 보정하고 규격화해 신경세포의 3차원 분포정보를 정확하게 얻을 수 있는 핵심 분석 기술을 개발했다고 8일 밝혔다.
이 기술은 실험자의 경험에 의존하던 기존 분석 방식의 문제점을 해결하고, 여러 개체에서 얻은 뇌 이미지를 표준적인 3차원 지도상에서 비교 분석할 수 있게 해준다.
이는 기존의 개체별 분석에서는 관측하기 힘든 뇌세포 간 상호 연결 형태의 정확한 공간적 분포를 발견할 수 있는 길을 열었다는 점에서 의미가 크다.
백 교수 연구팀은 동교 생명과학과 이승희 교수팀과의 협력 연구를 통해 실험에서 얻어진 쥐의 뇌 절편 데이터를 분석했다.
개발한 기술을 적용한 결과 시각시스템의 초기구조인 외측 슬상핵(Lateral geniculate nucleus)과 시각피질(Visual cortex) 사이의 정확한 연결 구조 분포를 측정할 수 있었다.
기존 분석 방식으로는 불가능했던 다중 개체로부터 얻어진 데이터의 표준화를 통해 뇌 전역에 걸친 신경세포의 연결성을 분석할 수 있음을 확인한 것이다.
백 교수 연구팀은 미국의 Allen Brain Atlas 프로젝트에서 제공한 쥐 두뇌의 3차원 표준 데이터에 기반해 SURF(Speeded Up Robust Feature Points) 특징점과 HOG(Histogram of Oriented Gradients descriptor) 형상 기술자를 이용, 데이터베이스와 비교하는 계산적인 분석 방법을 사용했다.
실험에서 얻은 뇌 이미지와 가장 잘 일치하는 데이터베이스의 3차원 위치를 100마이크로미터(μm), 1도 이내의 오차로 찾아낼 수 있었다.
연구팀은 이를 통해 각 2차원 뇌 이미지의 위치 정보를 3차원 공간상의 위치로 정확히 계산하고, 여러 개체에서 얻어진 신경 세포의 위치를 동일한 3차원 공간에 투영해 정확하게 분석할 수 있음을 확인했다.
이 기술을 활용하면 다양한 기법으로 생성된 뇌 슬라이스 이미지를 이용해 신경세포의 3차원 위치를 뇌 전체에서 자동적으로 계산할 수 있어 기존의 방법으로는 분석하기 어려운 수천~수만 개의 신경세포들의 정확한 뇌 내 분포 위치 및 상대적 공간 배열을 한번에 분석하는 것이 가능하다.
또 신경세포들의 연결성을 표준적으로 보정된 3차원 공간에서 표현할 수 있어 특정 뇌 영역 간의 연결은 물론 뇌 전역의 네트워크 분포를 여러 개체의 데이터를 사용해 동시분석도 가능해 기존 방식의 동물실험 분석에서 요구되던 시간과 비용을 크게 줄일 수 있을 것으로 기대된다.
백 교수는 "이번 연구를 통해 개발된 기술은 형광 뇌 이미지를 이용하는 모든 연구에 바로 적용할 수 있을 뿐만 아니라 그 밖에 다양한 종류의 이미지 데이터에도 광범위하게 적용 가능하다ˮ며 "향후 쥐의 뇌 슬라이스를 이용하는 다양한 분석에 표준적인 기법으로 자리잡을 수 있을 것으로 기대된다ˮ고 말했다.
이번 연구는 한국연구재단의 이공분야기초연구사업 및 원천기술개발사업, KAIST의 모험연구사업의 지원을 받아 수행됐다.
KAIST 뇌인지공학프로그램 최우철 박사과정과 송준호 연구원이 공동 제1 저자로 참여한 이번 연구 결과는 국제 학술지 `셀(cell)'의 온라인 자매지 `셀 리포츠(Cell Reports)'에 지난 5월 26일 게재됐다.
memory444444@nate.com
Copyright ⓒ 뉴스1. All rights reserved. 무단 전재 및 재배포, AI학습 이용금지.









